公共卫生学术热点追踪
Nature子刊:微生物所王军研究组结合三代测序合作解析肠道菌群结构变异和功能
近十年来,肠道微生物组已成为生命科学研究领域的热点,但目前大部分研究都集中在使用二代测序技术进行物种和功能的解析,宏基因组的拼接质量不高并且很难实现菌株水平的功能差异分析。
近日,中科院微生物研究所王军课题组和中科院动物研究所宋默识课题组合作,在 Nature 子刊 Nature Communications 上发表了题为:Short- and long-read metagenomics expand individualized structural variations in gut microbiomes 的研究论文。
该研究建立了牛津纳米孔三代测序和 Illumina 二代测序数据混合组装和后续分析流程。在 mock community 上的验证表明,三代和二代测序数据的混合组装从完整度、准确程度以及编码密度方面均比单纯二代或者三代测序组装更有优势。

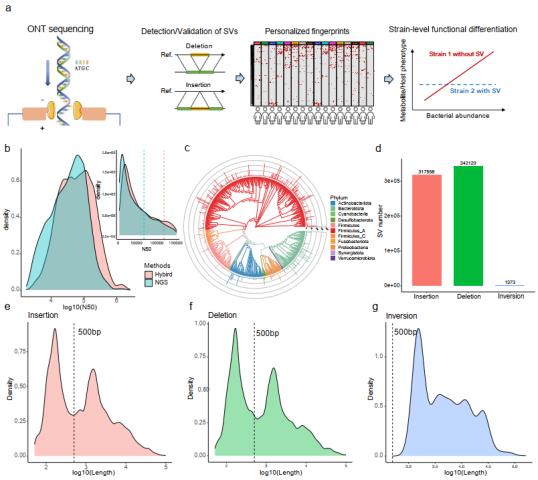
图1 本研究的技术路线(a),利用三代测序进行SV的深度解析,以及横断面/时间序列中SV的组成、动态分析,最终进行SV对代谢功能的影响判定。(b) 混合组装能够有效提高N50,并组装出大量的基因组(c),其中发现更多的insertion、deletion和inversion。
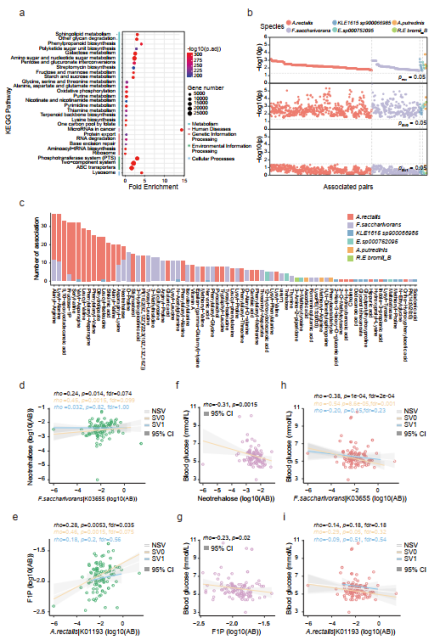
图2 肠道微生物中与SVs相关的功能研究结果。(a,b,c) SV影响基因富集结果; (d-i) SV影响单菌种内不同菌株与代谢产物以及血糖的关联。
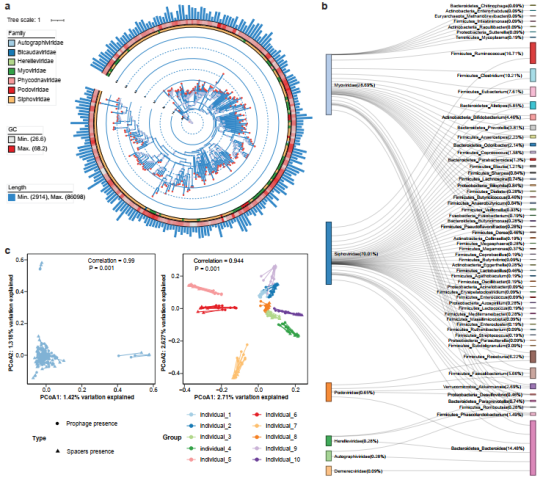
图3 肠道菌群汇中与病毒和CRISPR相关的研究结果
该研究基于三代ONT序列,提高了宏基因组装的质量、结构变异(Structural Variation,SV)的发现能力,发现了大量包括插入突变和基因倒位在内的结构变异对于菌株水平上基因功能的影响,以及噬菌体、CRISPR-spacer等系统的深度挖掘。这项研究是课题组利用三代纳米孔测序技术解析肠道病毒组(Cao et al., Medicine in Microecology, 2020, 4:100012; Cao et al. Gut Microbes, 2021, 13),近期发表的真菌组分析方法(Lu et al, Molecular Ecology, doi:10.1111/mec.16534)和靶向RNA检测病原微生物(Zhao et al., Advanced Science, 2021, 8, 2102593)之后,在利用三代纳米孔测序技术探索肠道微生物研究领域的新进展。这一发现,对未来更精细的精准医学领域的发展提供了理论启发。
(来源:中国生物技术网)
原文出处:Chen L, Zhao N, Cao J, Liu X, Xu J, Ma Y, Yu Y, Zhang X, Zhang W, Guan X, Yu X, Liu Z, Fan Y, Wang Y, Liang F, Wang D, Zhao L, Song M, Wang J. Short- and long-read metagenomics expand individualized structural variations in gut microbiomes. Nat Commun. 2022 Jun 8;13(1):3175. doi: 10.1038/s41467-022-30857-9. PMID: 35676264; PMCID: PMC9177567.
链接:https://pubmed.ncbi.nlm.nih.gov/35676264/
