公共卫生学术热点追踪
Cell Metab | 预先锻炼可启动信号以提高胰岛素抵抗个体对胰岛素的敏感性
胰岛素抵抗主要发生在骨骼肌、肝脏和脂肪组织,是与一系列胰岛素抵抗相关的代谢疾病如2型糖尿病(T2D)和心血管疾病(CVD)最早显露的缺陷之一,但人们对其确切机制仍缺乏了解。最近的研究表明胰岛素可调节数百个细胞上的数千个蛋白质的磷酸化位点,但其中只有10%属于经典的胰岛素信号通路,而大多数磷酸化位点的上游调控和下游功能未知[1-3]。
近日,来自丹麦哥本哈根大学的Jørgen Frank Pind Wojtaszewski团队在Cell Metabolism杂志上发表了一篇题为Personalized phosphoproteomics of skeletal muscle insulin resistance and exercise links MINDY1 to insulin action的文章,他们检测了胰岛素抵抗(IR)和胰岛素敏感(IS)男性对运动和胰岛素的反应时的磷酸化蛋白质组,发现在IR个体中预先锻炼可以通过在胰岛素输注之前启动部分信号(如AKT底物MINDY1 S441)来提高胰岛素敏感性,并证实MINDY1敲低可增强大鼠成肌细胞中胰岛素刺激的葡萄糖摄取,将MINDY1确定为胰岛素作用的调节剂。

该团队设计了一个实验流程检测预先运动对IR和IS个体代谢和分子差异的影响,简而言之,受试者先完成1小时单腿膝伸肌运动,恢复4小时后对运动和非运动腿股外侧肌进行活检,随后进行120分钟的高胰岛素-正常血糖钳夹,并在不同时间节点对每条腿进行活检,观察到预先锻炼可部分抵消骨骼肌胰岛素抵抗的影响。蛋白质组分析结果显示221种蛋白在IS和IR受试者中有差异,其中90种与腿部葡萄糖摄取(leg glucose uptake, LGU)相关的蛋白并不属于目前已知的胰岛素信号网络。由于胰岛素反应性LGU是一个动态过程,IS和IR之间的差异可能是由肌肉细胞内信号传导的变化驱动的,于是该团队进一步检测了磷酸化蛋白质组,胰岛素参与调节的磷酸蛋白质组富含参与典型胰岛素信号转导的激酶如AKT、mTOR、p70S6K、SGK和p90RSK,且IR中有缺陷的胰岛素反应位点富含mTOR底物如PATL1 S179、S184,MAF1 S75、AMPKa2 S377和MKNK2 S74。
此外,磷酸化蛋白质组中与已发表的与T2D、空腹血糖、糖化血红蛋白(HbA1c)、空腹胰岛素或糖尿病相关遗传变异最接近的17种蛋白质上的32个位点与具有胰岛素敏感性的受试者未运动腿中的LGU相关。其中包括在胰岛素信号传导(IRS1和IRS2)和代谢(PFKM、AMPD3和ACSL1)中具有已知作用的蛋白质,还有一些参与细胞信号传导但尚未在胰岛素反应中表征的蛋白质(MARK3、ARHGAP1、MINDY1和HBS1L),其中,MINDY1 (FAM63A) S441在IR和IS个体之间存在差异,目前还未知MINDY1 S441激酶,但他们观察到MINDY1 S441磷酸化能被已发表的磷酸化蛋白质组学资源中的5种不同AKT抑制剂消除,且AKT1、p70S6K和AKT2均可以不同程度地磷酸化该残基。AKT和p70S6K均被胰岛素强力激活且具有相似的共有基序,由于p70S6K位于mTORC1的下游,因此可以通过mTOR抑制剂加以区分。于是他们用rapamycin或rapalink-1处理HEK293细胞,发现MINDY1 S441磷酸化并不受影响,提示MINDY1 S441是AKT的直接底物。
最后,为了进一步探索MINDY1在胰岛素刺激的葡萄糖摄取中的潜在功能作用,他们敲低了胰岛素反应性大鼠L6(骨骼肌成肌细胞)中的Mindy1,其敲除大概率不会改变基态葡萄糖摄取,但会导致胰岛素刺激下对葡萄糖摄取的作用增强1.7倍。这与之前的观察结果一致,即MINDY1错义变异可能会对T2D具有保护作用。
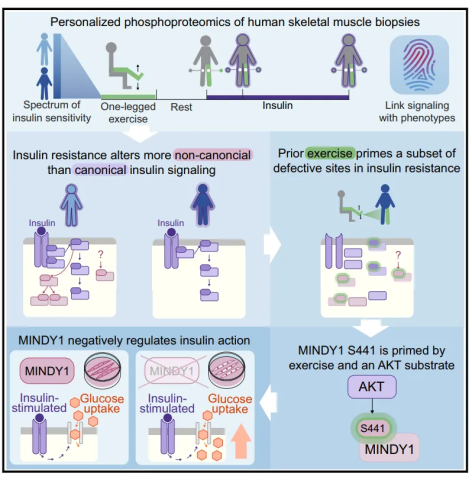
综上,该团队通过蛋白组学、磷酸蛋白组探索预先运动如何通过信号传导介导胰岛素敏感性,以及AKT下游靶标MINDY1对胰岛素介导的葡萄糖摄取的负调节作用,考虑到MINDY1功能丧失的错义变异对T2D具有保护作用[4, 5],且观察到Mindy1的敲低会增加胰岛素敏感性,AKT下游的MINDY1 S441磷酸化可能以抑制方式发挥作用,因此,未来研究磷酸化和错义变异如何改变MINDY1功能将有助于阐明其在胰岛素作用中的作用。
(来源:BioArtMED)
参考文献:
1. Kruger, M., Kratchmarova, I., Blagoev, B., Tseng, Y.-H., Kahn, C.R., and €Mann, M. (2008). Dissection of the insulin signaling pathway via quantitative phosphoproteomics. Proc. Natl. Acad. Sci. USA 105, 2451–2456. https://doi.org/10.1073/pnas.0711713105.
2. Nagao, H., Cai, W., Wewer Albrechtsen, N.J., Steger, M., Batista, T.M., Pan, H., Dreyfuss, J.M., Mann, M., and Kahn, C.R. (2021). Distinct signaling by insulin and IGF-1 receptors and their extra- and intracellular domains. Proc. Natl. Acad. Sci. USA 118, e2019474118. https://doi.org/10.1073/pnas.2019474118.
3. Needham, E.J., Parker, B.L., Burykin, T., James, D.E., and Humphrey, S.J.(2019). Illuminating the dark phosphoproteome. Sci. Signal. 12, eaau8645. https://doi.org/10.1126/scisignal.aau8645.
4. Type 2 Diabetes Knowledge Portal (2024). MINDY1 Region page. RRID:SCR_003743. type2diabetesgenetics.org.
5. Costanzo, M.C., von Grotthuss, M., Massung, J., Jang, D., Caulkins, L.,Koesterer, R., Gilbert, C., Welch, R.P., Kudtarkar, P., Hoang, Q., et al.(2023). The Type 2 Diabetes Knowledge Portal: An open access genetic resource dedicated to type 2 diabetes and related traits. Cell Metab. 35,695–710.e6. https://doi.org/10.1016/j.cmet.2023.03.001.
