公共卫生学术热点追踪
Cell:肠道菌群稳定的儿童,身高发育更好
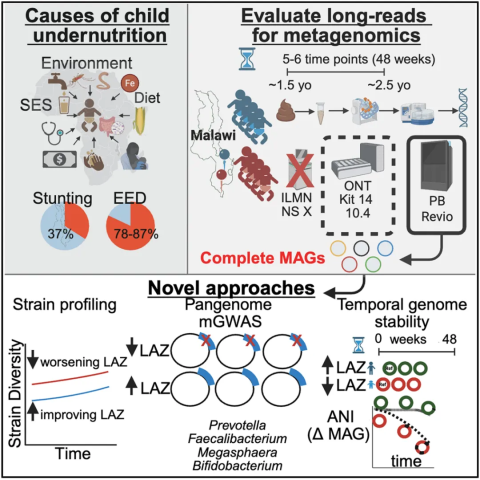
营养不良是 5 岁以下儿童死亡的主要原因。在全世界范围内,有近 1.5 亿的 5 岁以下儿童因缺乏营养而发育迟缓(身高低于同龄标准)。生命最初 1000 天内的营养不良尤其有害,会导致不可逆的长期认知和发育损害,以及将来的学业成绩不佳、经济劣势等。东南亚和撒哈拉以南的非洲的营养不良水平最高,其中,马拉维共和国的发育迟缓问题最为突出,发生率高达 37%。
饮食不足是儿童发育迟缓的主要原因,但多年前的研究就已发现,肠道微生物的功能失调在其中也发挥着重要作用。然而,传统的微生物组研究方法缺乏分辨率,难以确定哪些微生物发挥作用。
2025 年 9 月 9 日,索尔克研究所、加州大学圣地亚哥分校、贝勒医学院、圣路易斯华盛顿大学等机构的研究人员合作,在国际顶尖学术期刊 Cell 上发表了题为:Culture-independent meta-pangenomics enabled by long-read metagenomics reveals associations with pediatric undernutrition 的研究论文。
该研究利用了长读长宏基因组测序技术,无需进行传统的微生物培养,直接对样本中的全部基因组物质进行测序分析,构建了高质量的儿童微生物基因组数据库,研究团队对其中的马拉维地区儿童的粪便样本进行了深入分析,揭示了肠道微生物组的组成和稳定性与儿童线性生长(身高方面的生长)之间的密切关联,揭示了肠道微生物群稳定的儿童表现出更好的身高生长趋势。
这项技术的优势在于其高分辨率,能够提供菌株级的微生物信息,并通过泛基因组分析和微生物全基因组关联分析(mGWAS),发现了与儿童营养不良相关的特定微生物基因特征。这为在资源有限地区开展实时微生物监测和健康干预提供了新的技术路径。

早在二十多年前,该论文的共同通讯作者 Mark J. Manary 就成为了在马拉维推广基于花生酱的治疗性食品以对抗严重急性营养不良的关键人物。马拉维位于是撒哈拉以南的非洲的一个国家,该国家有高达 37%的儿童发育迟缓。他研发并临床测试了这种高热量、营养丰富的糊状食品,自其被采纳为全球治疗严重急性营养不良的标准疗法以来,已拯救了成千上万人的生命。
然而,曾患上严重急性营养不良的儿童,即便最终活下来,也常常面临新陈代谢、骨骼生长、免疫功能和大脑发育方面的持续挑战。因为仅仅提供食物让他们摄取足够的营养来恢复健康,这本身并不足以帮助他们成长和茁壮。
营养不良除了影响代谢、生长、免疫功能和大脑发育外,还会导致肠道微生物群失衡,减少有益微生物,增加致病微生物。研究人员推测,改善营养不良儿童的健康状况可能在于了解由数成百上千种细菌组成的肠道环境的变化。
为了弄清楚与儿童生长相关的微生物模式,研究团队构建了首个儿童微生物基因组数据库,其中包含来自马拉维的 8 名幼儿在 11 个月内的采集的粪便样本中 986 种微生物的完整基因图谱。这 8 名幼儿此前曾参与一项临床试验,该试验旨在测试以豆类为基础的辅食对减轻或逆转环境性肠病和生长不良的效果。
参与研究的幼儿的年龄在 12-24 个月之间,他们的年龄别身高 Z 评分(LAZ)要么在上升,要么在下降。LAZ 是评估儿童生长发育的重要指标,通过比较儿童身高与同年龄、同性别群体的平均值,计算标准差(Z 值)来判断其发育状况。
研究团队发现,肠道微生物基因组保持稳定的儿童(肠道微生物群没有发生剧烈变化)的生长状况,要优于肠道微生物基因组不稳定的儿童,这表明肠道微生物的稳定性可能有助于儿童的生长,监测此类变化或许可以用来评估肠道健康状况。
该研究采用了一种名为长读长测序的现代基因测序技术,来重建完整的微生物泛基因组,泛基因组包含了某一微生物物种所有成员的遗传物质。这种方法捕获的完整微生物群基因组数量是传统方法的 50 倍,并为高危儿童(易患发育迟缓和急性营养不良)体内微生物群提供了更全面的遗传视角。
具体来说,研究团队推测,通过长读长 DNA 测序恢复的完整宏基因组组装基因组(cMAG)能够实现泛基因组和微生物基因组范围关联研究(GWAS)分析,从而识别与儿童线性生长相关的微生物遗传关联。长读长方法每千兆碱基对(Gbp)产生的 cMAG 数量是短读长方法的 44-64 倍,其中 PacBio 测序技术产生的组装结果最准确且成本效益最高。
在马拉维的一个纵向儿科队列中,研究团队从 47 个样本中生成了 986 个微生物的完整或近乎完整的 cMAG(839 个为环状闭合的高质量基因组),并将此数据库应用于 210 个样本的扩展集。机器学习识别出了与线性生长相关的微生物物种。泛基因组分析揭示了微生物遗传与线性生长的关联,而基因组不稳定性与 LAZ 评分下降相关。这一资源展示了将 cMAG 与健康轨迹进行比较的强大之处,并为微生物组关联研究确立了新标准。
研究团队表示,发育迟缓和急性营养不良是由复杂多样的潜在过程导致的,可以通过简单的身体测量指标来定义。提高识别微生物群、它们的变化以及作用的分辨率和准确性,或许能让我们了解营养不良中那些原本无法测量的方面,帮助揭示肠道微生物群在其中所发挥的作用。
该技术使得构建这项研究中全新的儿童微生物基因组文库成为可能。研究团队优化了长读长测序工作流程,其他研究人员可以对其进行调整,以构建适用于各种应用的基因组文库,并且这种工作流程适合在难以到达的偏远地区开展研究的实验室使用。
研究团队表示,在偏远的野外分子实验室中应用该研究开发的基因组测序和泛基因组方法时,不仅能实时了解大流行病监测、抗生素耐药性和传染病的情况,还能了解农业生产力、环境监测和生物多样性保护方面的信息。总的来说,这是一项强大的技术进步,拓展了基因组学的研究范围,并为该领域的科学研究树立了新标准。
(来源:生物世界)
原文出处:Minich JJ, Allsing N, Din MO, Tisza MJ, Maleta K, McDonald D, Hartwick N, Mamerto A, Brennan C, Hansen L, Shaffer J, Murray ER, Duong T, Knight R, Stephenson K, Manary MJ, Michael TP. Culture-independent meta-pangenomics enabled by long-read metagenomics reveals associations with pediatric undernutrition. Cell. 2025 Sep 3:S0092-8674(25)00975-4. doi: 10.1016/j.cell.2025.08.020. Epub ahead of print. PMID: 40930091.
